#@title Importar librerías
#importar librerías necesarias
import random
from random import randint
import numpy as np
import pandas as pd
from tqdm.notebook import tqdm
from matplotlib import pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.datasets import fetch_openml
from sklearn.metrics import classification_report, confusion_matrix
import seaborn as sns
import random
import tensorflow as tf
import tensorflow.keras as keras
from tensorflow.keras.layers import Dense, Dropout, Activation, Input
from tensorflow.keras.optimizers import SGD
from tensorflow.keras.utils import to_categorical
from tensorflow.keras.datasets import mnistImplementado una Red Neuronal Convolucional
Última actualización 09/05/2025#@title Funciones complementarias
def plot_samples_dataset(X, y):
# Convertir las etiquetas a enteros
y = y.astype(int)
# Crear la grilla de 4x4
fig, axes = plt.subplots(4, 4, figsize=(6, 6))
fig.suptitle('Grilla de imágenes del dataset MNIST')
# Iterar para mostrar las primeras 16 imágenes con sus etiquetas
for i, ax in enumerate(axes.flat):
img = X.iloc[i].values.reshape(28, 28)
label = y[i]
ax.imshow(img, cmap='gray')
ax.set_title(f'Label: {label}')
ax.axis('off')
plt.show()
def plot_curva_aprendizaje(mlp):
plt.figure(figsize=(8, 5))
plt.plot(mlp.loss_curve_, marker='o')
plt.title('Pérdida durante el entrenamiento del MLP por iteración')
plt.xlabel('Iteración')
plt.ylabel('Pérdida (Loss)')
plt.grid()
plt.show()
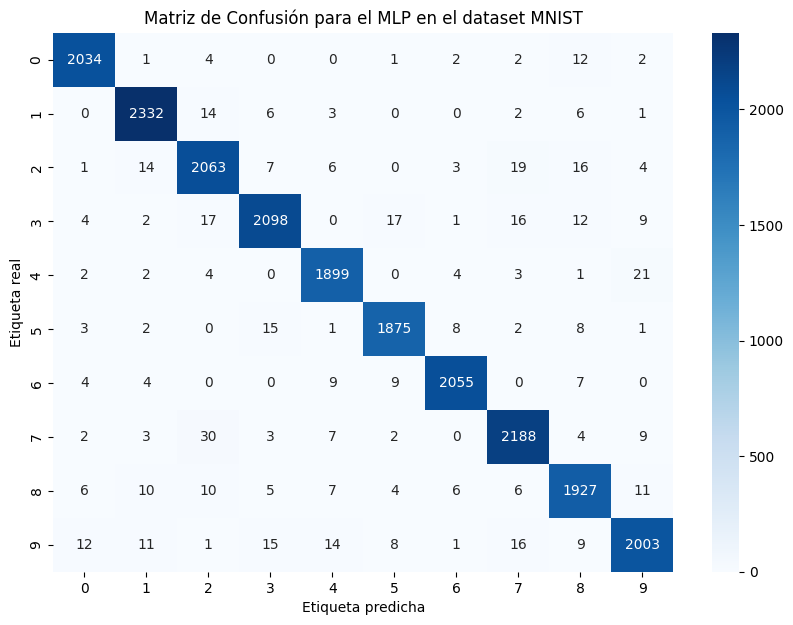
def plot_matriz_confusion(cm):
# Visualizar la matriz de confusión usando Seaborn
plt.figure(figsize=(10, 7))
sns.heatmap(cm, annot=True, fmt='d', cmap='Blues', xticklabels=range(10), yticklabels=range(10))
plt.xlabel('Etiqueta predicha')
plt.ylabel('Etiqueta real')
plt.title('Matriz de Confusión para el MLP en el dataset MNIST')
plt.show()
def encontrar_dim_imagen(n_neurons):
"""
Encuentra la mejor forma cuadrada (filas, columnas) para una cantidad dada de neuronas.
"""
side_length = int(np.sqrt(n_neurons)) # Calcular la raíz cuadrada del número de neuronas
if side_length * side_length == n_neurons:
return (side_length, side_length) # Si es un cuadrado perfecto
else:
# Si no es un cuadrado perfecto, buscamos la mejor aproximación (filas, columnas)
for i in range(side_length, 0, -1):
if n_neurons % i == 0:
return (i, n_neurons // i) # Devolver filas y columnas
return (n_neurons, 1) # Si no encuentra, retornar en forma de vector (n_neurons, 1)
def plot_loss_historia_keras(history):
# Graficar el histórico de pérdida durante el entrenamiento
plt.plot(history.history['loss'], label='Pérdida de Entrenamiento')
plt.plot(history.history['val_loss'], label='Pérdida de Validación')
plt.title('Pérdida durante el Entrenamiento')
plt.xlabel('Época')
plt.ylabel('Pérdida')
plt.legend()
plt.show()
def plot_acc_historia_keras(history):
# Graficar la precisión durante el entrenamiento
plt.plot(history.history['accuracy'], label='Precisión de Entrenamiento')
plt.plot(history.history['val_accuracy'], label='Precisión de Validación')
plt.title('Precisión durante el Entrenamiento')
plt.xlabel('Época')
plt.ylabel('Precisión')
plt.legend()
plt.show()
def visualizacion_filtros_cnn_keras(model):
# Obtener las capas convolucionales del modelo
conv_layers = [layer for layer in model.layers if 'conv' in layer.name]
# Definir la figura con una fila por capa convolucional y varias columnas (5 filtros al azar por capa)
fig, axes = plt.subplots(len(conv_layers), 5, figsize=(10, len(conv_layers) * 3))
# Recorrer cada capa convolucional y sus pesos
for layer_index, (layer, ax_row) in enumerate(zip(conv_layers, axes)):
# Obtener los pesos de la capa (solo el primer tensor, ignorar bias)
layer_weights = layer.get_weights()[0] # shape: (filter_height, filter_width, input_channels, num_filters)
# Seleccionar 5 filtros de la capa actual
num_filters = layer_weights.shape[-1]
random_filters = random.sample(range(num_filters), 5)
# Obtener los límites para la normalización de las imágenes
vmin, vmax = layer_weights.min(), layer_weights.max()
# Dibujar cada filtro seleccionado
for filter_index, ax in zip(random_filters, ax_row):
# Extraer el filtro correspondiente (shape: filter_height, filter_width, input_channels)
filter_weights = layer_weights[..., filter_index]
# Promediar los canales para visualizar como imagen en escala de grises
if filter_weights.shape[-1] > 1:
filter_weights = np.mean(filter_weights, axis=-1) # Promedio sobre los canales de entrada
# Dibujar la imagen del filtro
ax.matshow(filter_weights, cmap=plt.cm.gray, vmin=0.5 * vmin, vmax=0.5 * vmax)
ax.set_xticks([])
ax.set_yticks([])
ax.set_title(f'Capa {layer_index + 1}, Filtro {filter_index}')
plt.suptitle('Visualización de Filtros de las Capas Convolucionales de Keras')
plt.tight_layout()
plt.show()
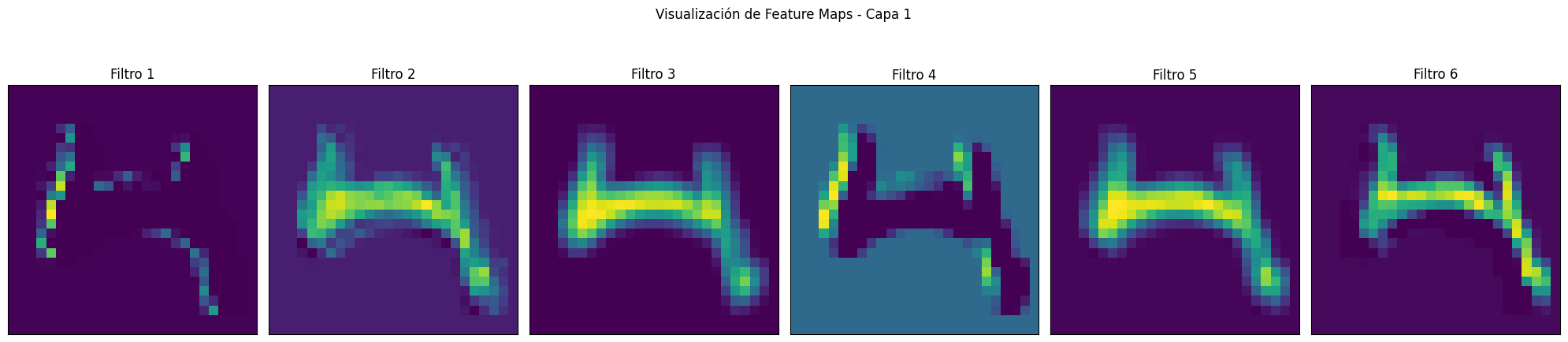
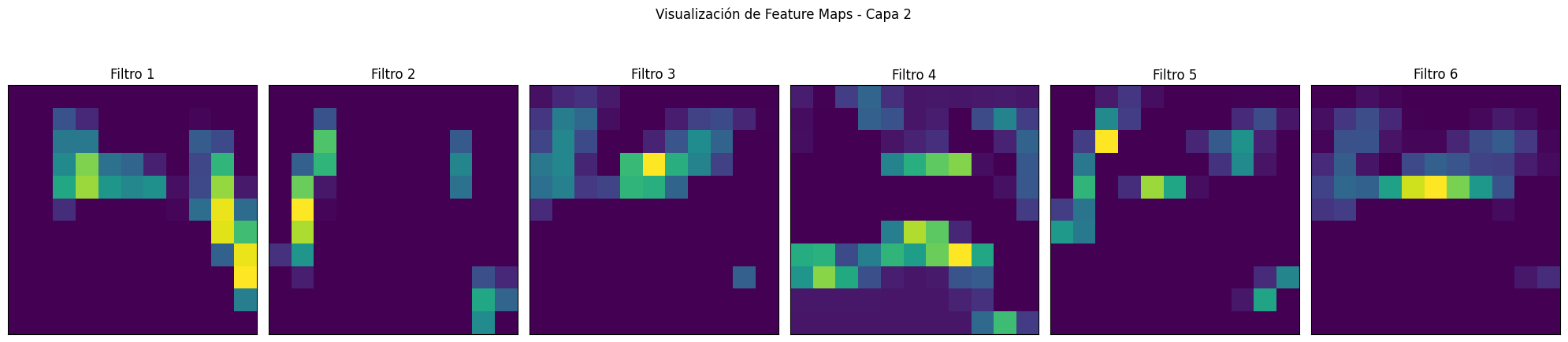
def visualizacion_feature_maps(model, image):
# Crear un nuevo modelo que toma la misma entrada pero cuya salida son los mapas de características de cada capa convolucional
layer_outputs = [layer.output for layer in model.layers if 'conv' in layer.name]
feature_map_model = tf.keras.Model(inputs=model.input, outputs=layer_outputs)
# Obtener los mapas de características al pasar la imagen a través del modelo
feature_maps = feature_map_model.predict(np.expand_dims(image, axis=0)) # Añadir batch dimension
# Recorrer cada capa convolucional y sus feature maps correspondientes
for layer_index, feature_map in enumerate(feature_maps):
# Número de filtros en la capa actual
num_filters = feature_map.shape[-1]
# Definir la figura con una fila por cada filtro (limitado a 6 para evitar gráficos muy grandes)
fig, axes = plt.subplots(1, min(6, num_filters), figsize=(20, 5))
# Mostrar cada filtro como imagen en escala de grises
for i in range(min(6, num_filters)): # Mostrar un máximo de 6 filtros
ax = axes[i]
# Extraer el feature map del filtro `i` y mostrarlo
ax.matshow(feature_map[0, :, :, i], cmap='viridis')
ax.set_xticks([])
ax.set_yticks([])
ax.set_title(f'Filtro {i+1}')
# Título de la capa y ajuste de la visualización
plt.suptitle(f'Visualización de Feature Maps - Capa {layer_index+1}')
plt.tight_layout()
plt.show()Dataset MNIST (Clasificación multiclase)
Trabajaremos con el clásico dataset de imágenes de digitos escritos a mano. Esta dataset puede ser descargado de diversas fuentes incluido sklearn.
# Cargar el dataset de MNIST
mnist = fetch_openml('mnist_784', version=1)
X, y = mnist['data'], mnist['target']# Visualizamos algunas imágenes
plot_samples_dataset(X, y)
Un breve vistazo del dataset nos indica que la mayoría de pixeles de la imágen estan en 0 y que los que tiene valor estan en 255. Esta escala, es los valores de intensidad en escala de grises. Además, vemos que la información esta organizada en un dataframe con 784 columnas lo que se traduce en una versión vectorizada de una imágen de 28x28 píxeles.
X.describe()| pixel1 | pixel2 | pixel3 | pixel4 | pixel5 | pixel6 | pixel7 | pixel8 | pixel9 | pixel10 | ... | pixel775 | pixel776 | pixel777 | pixel778 | pixel779 | pixel780 | pixel781 | pixel782 | pixel783 | pixel784 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | 70000.0 | ... | 70000.000000 | 70000.000000 | 70000.000000 | 70000.000000 | 70000.000000 | 70000.000000 | 70000.0 | 70000.0 | 70000.0 | 70000.0 |
| mean | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.197414 | 0.099543 | 0.046629 | 0.016614 | 0.012957 | 0.001714 | 0.0 | 0.0 | 0.0 | 0.0 |
| std | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 5.991206 | 4.256304 | 2.783732 | 1.561822 | 1.553796 | 0.320889 | 0.0 | 0.0 | 0.0 | 0.0 |
| min | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.0 | 0.0 | 0.0 | 0.0 |
| 25% | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.0 | 0.0 | 0.0 | 0.0 |
| 50% | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.0 | 0.0 | 0.0 | 0.0 |
| 75% | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.0 | 0.0 | 0.0 | 0.0 |
| max | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 254.000000 | 254.000000 | 253.000000 | 253.000000 | 254.000000 | 62.000000 | 0.0 | 0.0 | 0.0 | 0.0 |
8 rows × 784 columns
Antes de hacer la división del conjunto de datos, podemos hacer una normalización de los valores de los píxeles para que esten entre 0-1, así es más fácil para la red optimizar la función de pérdida.
# Normalización
X = X / 255.# usar train test split para dividir los datos X y y
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size=0.30, random_state=42)
print('Dimensión X_train: {}'.format(X_train.shape))
print('Dimensión X_test: {}'.format(X_test.shape))Dimensión X_train: (49000, 784)
Dimensión X_test: (21000, 784)La entrada a una red CNN es diferente que las entradas para un MLP. En primera instancia debemos redimensionar nuestro vector de 784 pixeles en un arreglo de 28x28, lo cual representa nuestra imagen de digitos. Por otro lado, en las CNN es obligatorio agregar una tercera dimensión a la matriz de entrada, ya que las imágenes se representa por medio de canales. Cuando son en blanco y negro, se dice que solo tienen un canal y cuando son a color (RGB) son 3 canales.
n_muestras_train = X_train.shape[0]
n_muestras_test = X_test.shape[0]
dim_imagen = (28, 28)
n_canales = 1
# Aplicar el reespectivo redimensionamiento
X_train = np.reshape(X_train, (n_muestras_train,
dim_imagen[1], dim_imagen[1],
n_canales))
X_test = np.reshape(X_test, (n_muestras_test,
dim_imagen[1], dim_imagen[1],
n_canales))
print('Dimensión X_train: {}'.format(X_train.shape))
print('Dimensión X_test: {}'.format(X_test.shape))Dimensión X_train: (49000, 28, 28, 1)
Dimensión X_test: (21000, 28, 28, 1)Clasificador CNN multiclase en keras
Componentes base complementarios
- Capas de Pooling
- Capas de normalización
1. Capas de Pooling
2. Capas de normalización
En nuestro ejemplo, no es necesario normalizar ya que nuestras entradas estan entre 0 y 1 y no tienen un alta complejidad. Pero en otros escenarios, con imágenes más complejas y en formato RGB la normalización se hace más común.
# Asumiendo que X_train y y_train ya están definidos como en el ejemplo anterior
# Preprocesar las etiquetas para que sean categóricas (one-hot encoding)
y_train_categorical = to_categorical(y_train)
y_train_categoricalarray([[1., 0., 0., ..., 0., 0., 0.],
[0., 0., 0., ..., 0., 0., 0.],
[0., 0., 0., ..., 0., 0., 0.],
...,
[0., 1., 0., ..., 0., 0., 0.],
[1., 0., 0., ..., 0., 0., 0.],
[1., 0., 0., ..., 0., 0., 0.]])# crear modelo usando el API funcional
def cnn_model(input_shape, num_classes):
# Definir la entrada
inputs = tf.keras.Input(shape=input_shape)
# Primera capa convolucional y de pooling
x = tf.keras.layers.Conv2D(32, kernel_size=(3, 3), activation="relu",
strides=(1, 1),
padding="valid")(inputs)
x = tf.keras.layers.MaxPooling2D(pool_size=(2, 2))(x)
# Segunda capa convolucional y de pooling
x = tf.keras.layers.Conv2D(64, kernel_size=(3, 3), activation="relu",
strides=(1, 1),
padding="valid")(x)
x = tf.keras.layers.MaxPooling2D(pool_size=(2, 2))(x)
# Aplanar y añadir Dropout
x = tf.keras.layers.Flatten()(x)
x = tf.keras.layers.Dropout(0.5)(x)
# Capa de salida
outputs = tf.keras.layers.Dense(num_classes, activation="softmax")(x)
# Crear el modelo usando la API funcional
model = tf.keras.Model(inputs=inputs, outputs=outputs, name='cnn_keras')
return model# Ahora se definen los input shape y el numero de clases
input_shape = (dim_imagen[0], dim_imagen[1], n_canales)
num_classes = 10
# Crear el modelo cnn
cnn_keras = cnn_model(input_shape, num_classes)
# Visualizar el resumen del modelo
cnn_keras.summary()Model: "cnn_keras"
┏━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━┓ ┃ Layer (type) ┃ Output Shape ┃ Param # ┃ ┡━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━┩ │ input_layer (InputLayer) │ (None, 28, 28, 1) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ conv2d (Conv2D) │ (None, 26, 26, 32) │ 320 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ max_pooling2d (MaxPooling2D) │ (None, 13, 13, 32) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ conv2d_1 (Conv2D) │ (None, 11, 11, 64) │ 18,496 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ max_pooling2d_1 (MaxPooling2D) │ (None, 5, 5, 64) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ flatten (Flatten) │ (None, 1600) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ dropout (Dropout) │ (None, 1600) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ dense (Dense) │ (None, 10) │ 16,010 │ └─────────────────────────────────┴────────────────────────┴───────────────┘
Total params: 34,826 (136.04 KB)
Trainable params: 34,826 (136.04 KB)
Non-trainable params: 0 (0.00 B)
# tambien es posible visualizar nuestra red en formato de grafo
tf.keras.utils.plot_model(cnn_keras, rankdir='LR',show_dtype=True)
# Compilar el modelo
cnn_keras.compile(loss='categorical_crossentropy',
optimizer=SGD(),
metrics=['accuracy'])
# Entrenar el modelo
history = cnn_keras.fit(X_train, y_train_categorical,
epochs=30,
batch_size=128,
validation_split=0.2,
verbose=1)Epoch 1/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 32s 103ms/step - accuracy: 0.2604 - loss: 2.1329 - val_accuracy: 0.7763 - val_loss: 0.8651 Epoch 2/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 40s 100ms/step - accuracy: 0.7170 - loss: 0.8741 - val_accuracy: 0.8841 - val_loss: 0.4328 Epoch 3/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 102ms/step - accuracy: 0.8282 - loss: 0.5398 - val_accuracy: 0.9079 - val_loss: 0.3207 Epoch 4/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 30s 98ms/step - accuracy: 0.8755 - loss: 0.4041 - val_accuracy: 0.9270 - val_loss: 0.2540 Epoch 5/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 100ms/step - accuracy: 0.8992 - loss: 0.3252 - val_accuracy: 0.9372 - val_loss: 0.2167 Epoch 6/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 101ms/step - accuracy: 0.9138 - loss: 0.2829 - val_accuracy: 0.9444 - val_loss: 0.1931 Epoch 7/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 101ms/step - accuracy: 0.9231 - loss: 0.2526 - val_accuracy: 0.9512 - val_loss: 0.1733 Epoch 8/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 99ms/step - accuracy: 0.9292 - loss: 0.2346 - val_accuracy: 0.9535 - val_loss: 0.1600 Epoch 9/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 30s 96ms/step - accuracy: 0.9350 - loss: 0.2114 - val_accuracy: 0.9570 - val_loss: 0.1478 Epoch 10/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 31s 100ms/step - accuracy: 0.9376 - loss: 0.2025 - val_accuracy: 0.9600 - val_loss: 0.1391 Epoch 11/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 102ms/step - accuracy: 0.9452 - loss: 0.1841 - val_accuracy: 0.9620 - val_loss: 0.1314 Epoch 12/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 40s 99ms/step - accuracy: 0.9466 - loss: 0.1766 - val_accuracy: 0.9627 - val_loss: 0.1269 Epoch 13/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 30s 99ms/step - accuracy: 0.9497 - loss: 0.1683 - val_accuracy: 0.9649 - val_loss: 0.1202 Epoch 14/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 43s 105ms/step - accuracy: 0.9497 - loss: 0.1671 - val_accuracy: 0.9664 - val_loss: 0.1161 Epoch 15/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 30s 96ms/step - accuracy: 0.9530 - loss: 0.1590 - val_accuracy: 0.9683 - val_loss: 0.1109 Epoch 16/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 99ms/step - accuracy: 0.9527 - loss: 0.1602 - val_accuracy: 0.9686 - val_loss: 0.1073 Epoch 17/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 99ms/step - accuracy: 0.9549 - loss: 0.1468 - val_accuracy: 0.9685 - val_loss: 0.1040 Epoch 18/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 102ms/step - accuracy: 0.9561 - loss: 0.1420 - val_accuracy: 0.9698 - val_loss: 0.1018 Epoch 19/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 39s 96ms/step - accuracy: 0.9587 - loss: 0.1382 - val_accuracy: 0.9705 - val_loss: 0.0985 Epoch 20/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 32s 103ms/step - accuracy: 0.9579 - loss: 0.1386 - val_accuracy: 0.9718 - val_loss: 0.0948 Epoch 21/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 40s 99ms/step - accuracy: 0.9611 - loss: 0.1306 - val_accuracy: 0.9710 - val_loss: 0.0932 Epoch 22/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 103ms/step - accuracy: 0.9593 - loss: 0.1307 - val_accuracy: 0.9719 - val_loss: 0.0905 Epoch 23/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 40s 99ms/step - accuracy: 0.9597 - loss: 0.1280 - val_accuracy: 0.9717 - val_loss: 0.0887 Epoch 24/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 30s 99ms/step - accuracy: 0.9629 - loss: 0.1202 - val_accuracy: 0.9738 - val_loss: 0.0870 Epoch 25/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 31s 100ms/step - accuracy: 0.9644 - loss: 0.1226 - val_accuracy: 0.9735 - val_loss: 0.0851 Epoch 26/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 42s 104ms/step - accuracy: 0.9641 - loss: 0.1221 - val_accuracy: 0.9745 - val_loss: 0.0839 Epoch 27/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 39s 99ms/step - accuracy: 0.9659 - loss: 0.1155 - val_accuracy: 0.9742 - val_loss: 0.0822 Epoch 28/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 99ms/step - accuracy: 0.9650 - loss: 0.1146 - val_accuracy: 0.9745 - val_loss: 0.0809 Epoch 29/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 41s 99ms/step - accuracy: 0.9648 - loss: 0.1158 - val_accuracy: 0.9746 - val_loss: 0.0815 Epoch 30/30 307/307 ━━━━━━━━━━━━━━━━━━━━ 31s 100ms/step - accuracy: 0.9655 - loss: 0.1112 - val_accuracy: 0.9764 - val_loss: 0.0780
plot_loss_historia_keras(history)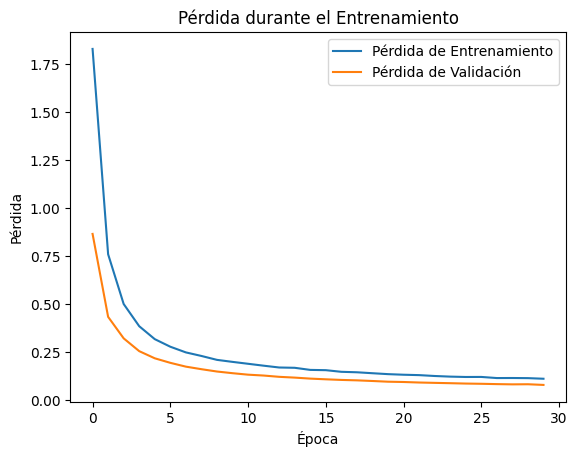
plot_acc_historia_keras(history)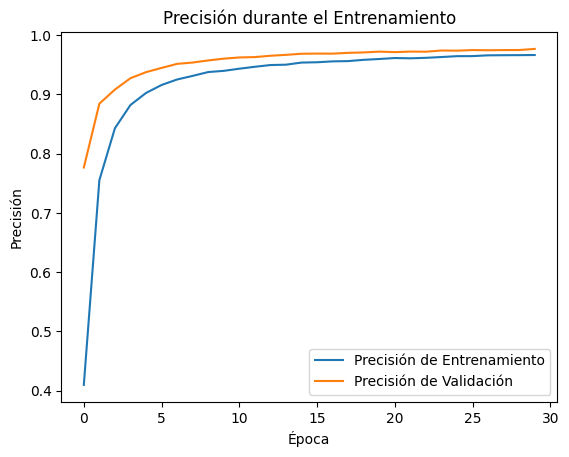
Evaluación completa
y_test_categorical = to_categorical(y_test)
score = cnn_keras.evaluate(X_test, y_test_categorical, batch_size=128)
score165/165 ━━━━━━━━━━━━━━━━━━━━ 4s 24ms/step - accuracy: 0.9740 - loss: 0.0853
[0.08383515477180481, 0.9749523997306824]# Realizar predicciones en el conjunto de prueba
y_pred = cnn_keras.predict(X_test)
# Convertir las predicciones en etiquetas (la clase con mayor probabilidad)
y_pred_classes = np.argmax(y_pred, axis=1)
y_true = y_test.values.astype(int) # Las etiquetas reales del conjunto de prueba657/657 ━━━━━━━━━━━━━━━━━━━━ 5s 8ms/step
# Generar el reporte de clasificación
print("Reporte de Clasificación para la red CNN en MNIST:\n")
print(classification_report(y_true, y_pred_classes))
# Crear la matriz de confusión
cm = confusion_matrix(y_true, y_pred_classes)
# Visualizar la matriz de confusión usando Seaborn
plot_matriz_confusion(cm)Reporte de Clasificación para la red CNN en MNIST:
precision recall f1-score support
0 0.98 0.99 0.99 2058
1 0.98 0.99 0.98 2364
2 0.96 0.97 0.96 2133
3 0.98 0.96 0.97 2176
4 0.98 0.98 0.98 1936
5 0.98 0.98 0.98 1915
6 0.99 0.98 0.99 2088
7 0.97 0.97 0.97 2248
8 0.96 0.97 0.96 1992
9 0.97 0.96 0.97 2090
accuracy 0.97 21000
macro avg 0.97 0.97 0.97 21000
weighted avg 0.97 0.97 0.97 21000
Visualización de filtros convolucionales
visualizacion_filtros_cnn_keras(cnn_keras)
Visualización de los features maps
visualizacion_feature_maps(cnn_keras, image=X_test[randint(0, 100),:,:,:])1/1 ━━━━━━━━━━━━━━━━━━━━ 0s 70ms/step